GUC
Bereitstellung von Daten aus der DiversityWorkbench für die GUC
Anmelden
Beim Start des Programms müssen zunächst die Login Daten eingegeben werden.
Danach startet das Programm mit dem Hauptfenster.
Bereitstellung von Daten aus der DiversityWorkbench für die GUC
Beim Start des Programms müssen zunächst die Login Daten eingegeben werden.

Danach startet das Programm mit dem Hauptfenster.
Das Setup für das Programm liegt bereit unter:
 Artcode: SQL-Server Funktionen überarbeitet und Aufruf des Transfers der Merkmale in Client integriert.
Artcode: SQL-Server Funktionen überarbeitet und Aufruf des Transfers der Merkmale in Client integriert.
sqlite.db im Userverzeichnis .../Users/[Login]/DiversityWorkbench/GUC/Artcode löschen oder umbenennen damit der Export korrekt arbeitet. Artcode: Neue Spalten für RL-A (Gefährdung nach Roter Liste Bayern 2024 - Alpen) und RL-HB
Artcode: Neue Spalten für RL-A (Gefährdung nach Roter Liste Bayern 2024 - Alpen) und RL-HB  .
.
sqlite.db im Userverzeichnis .../Users/[Login]/DiversityWorkbench/GUC/Artcode löschen oder umbenennen damit die Spalten neu erzeugt werden. Bugfix Export nach SQLite
Bugfix Export nach SQLite Bugfix Export nach SQLite
Bugfix Export nach SQLite Bugfix Export
Bugfix Export Bugfixes creating the SQLite database
Bugfixes creating the SQLite database Bugfix Login
Bugfix Login Die Seiten des Manuals werden noch nicht erkannt
Die Seiten des Manuals werden noch nicht erkannt Anpassung der Prozedur zum Export der Daten
Anpassung der Prozedur zum Export der DatenFor users that need more detailed information about the software including database design etc.
Die Hauptfunktion zum Übertrag der Daten startet eine Prozedur (TransferToGUC) in der Datenbank. Diese ruft nacheinander andere Prozeduren auf um die Daten in die entsprechenden Tabellen zu transferieren.
graph TD; Tnt[<i class="fa-fw fas fa-server"></i> Server<br>TNT] -->DTN_Plants(<i class="fa-fw fas fa-database"></i> Database<br>DTN_Plants)-->DTN_Plants_Bfl(<i class="fa-fw fas fa-lock"></i> Projekt<br>BFLnames) Bfl[<i class="fa-fw fas fa-server"></i> Server<br>BFL] -->DC_Bfl(<i class="fa-fw fas fa-database"></i> Database<br>DC_Bayernflora)-->DC_Bfl_3(<i class="fa-fw fas fa-lock"></i> Projekt<br>BFLportal03coll) DC_Bfl_3-->DC_Bfl_Cache(<i class="fa-fw fas fa-database"></i> Database<br>DC_BayernfloraCache) DTN_Plants_Bfl-->DC_Bfl_Cache(<i class="fa-fw fas fa-database"></i> Database<br>DC_BayernfloraCache) DC_Bfl_Cache-->DC_Bfl_Guc_Cache(<i class="fa-fw fas fa-database"></i> Database<br>DC_BayernfloraGucCache)
Unten findet sich eine Übersicht der einzelnen Schritte.
--- title: PROCEDURE TransferToGUC --- graph TD; TransferToGUC[<i class="fa-fw fas fa-arrows-rotate"></i> Hauptprozedur<br>zum Übertrag] -->Festhalten_Zeitpunkts(<i class="fa-fw fas fa-stopwatch"></i> Festhalten<br>des Zeitpunkts)-->Festhalten_Zeitpunkte(<i class="fa-fw fas fa-stopwatch"></i> Festhalten<br>des Zeitpunkts<br>aller Schritte<br>→Tabelle _Log)-->fundort_datum[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen der Daten in<br>_fundort_letzte_Aend...<br>→Tabelle _Log]-->Alle_Zeitpunkte[<i class="fa-fw fas fa-stopwatch"></i> ...<br>→Tabelle _Log] Festhalten_Zeitpunkts-->LoeschenTabellen(<i class="fa-fw fas fa-trash"></i> Löschen<br>der Tabellen:<br>_Status, <br>NACHWEIS, <br>FUNDORT, <br>SAMMLER) -->Status[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen der Daten<br>in _Status] LoeschenTabellen-->_Identification[<i class="fa-fw fas fa-table"></i> Tabelle<br>_Identification] -->Loeschen_Identification(<i class="fa-fw fas fa-trash"></i> Löschen<br>der Daten)-->Einlesen_Identification[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten ]-->Loeschen_Identification_ohne_artencode(<i class="fa-fw fas fa-trash"></i> Entfernung<br>von Daten<br>die keinen<br>artencode haben) Loeschen_Identification_ohne_artencode-->Einlesen_Nachweis LoeschenTabellen-->Nachweis[<i class="fa-fw fas fa-table"></i> Tabelle<br>NACHWEIS]-->Einlesen_Nachweis[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten]-->Loeschen_Nachweis_ohne_Jahr(<i class="fa-fw fas fa-trash"></i>Entfernung<br>von Daten<br>die kein<br>Jahr haben)-->Eintag_Nachweis_Status(<i class="fa-fw fas fa-pen"></i> Eintrag<br>des Status)-->Eintag_Nachweis_quellentyp(<i class="fa-fw fas fa-pen"></i> Eintrag<br>des Quellentyp)-->Eintag_Nachweis_verbleib_nachname(<i class="fa-fw fas fa-pen"></i> Eintrag<br>verbleib_nachname)-->Eintag_Nachweis_projekt(<i class="fa-fw fas fa-pen"></i> Eintrag<br>projekt)-->Eintag_Nachweis_VERORTUNG_PUNKT_WKT(<i class="fa-fw fas fa-pen"></i> Eintrag<br>VERORTUNG_PUNKT_WKT)-->Eintag_Nachweis_status_code_lfu(<i class="fa-fw fas fa-pen"></i> Eintrag<br>status_code_lfu)-->Export_Nachweis(<i class="fa-fw fas fa-download"></i>) Status-->Eintag_Nachweis_Status LoeschenTabellen-->FUNDORT[<i class="fa-fw fas fa-table"></i> Tabelle<br>FUNDORT]-->Einlesen_FUNDORT[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten]-->Eintag_FUNDORT_tk(<i class="fa-fw fas fa-pen"></i> Eintrag<br>TK, Quadrant, ...)-->Eintag_FUNDORT_toponym(<i class="fa-fw fas fa-pen"></i> Eintrag<br>toponym)-->Eintag_FUNDORT_Hoehe(<i class="fa-fw fas fa-pen"></i> Eintrag<br>Hoehe)-->Eintag_FUNDORT_lebensraum(<i class="fa-fw fas fa-pen"></i> Eintrag<br>lebensraum)-->Eintag_FUNDORT_WGS84(<i class="fa-fw fas fa-pen"></i> Eintrag<br>WGS84)-->Eintag_FUNDORT_naturraum(<i class="fa-fw fas fa-pen"></i> Eintrag<br>naturraum)-->Eintag_FUNDORT_landkreis(<i class="fa-fw fas fa-pen"></i> Eintrag<br>landkreis)-->Eintag_FUNDORT_naturraum_code_lfu(<i class="fa-fw fas fa-pen"></i> Eintrag<br>naturraum_code_lfu)-->Eintag_FUNDORT_landkreis_code_lfu(<i class="fa-fw fas fa-pen"></i> Eintrag<br>landkreis_code_lfu)-->Export_Fundort(<i class="fa-fw fas fa-download"></i>) LoeschenTabellen-->SAMMLER[<i class="fa-fw fas fa-table"></i> Tabelle<br>SAMMLER]-->Einlesen_SAMMLER[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten]-->Export_Sammler(<i class="fa-fw fas fa-download"></i>)
---
title: PROCEDURE TransferToGUC
---
graph TD;
1{ } -->|1| Clear[Clear the export tables]
1 -->|2| Status[Tabelle <br><u>_Status</u><br> füllen]
1 --> |3|Identification[Tabelle <br>_Identification<br> füllen]
1 --> |4|FehlendeTaxa[Daten ohne Code aus <br>_Identification<br> entfernen]
1 --> |5|NACHWEIS[Tabelle <br>NACHWEIS<br> füllen]
FehlendeTaxa --> Identification
Identification --> NACHWEIS
Status --> EintragStatus
NACHWEIS --> Jahresangaben(In NACHWEIS:<br>Entfernen der Daten<br>ohne Jahresangaben)
Jahresangaben --> EintragStatus(In NACHWEIS<br>Eintrag des Status)
EintragStatus --> Quellentyp(In NACHWEIS<br>Eintrag des Quellentyp)
Quellentyp --> verbleib_nachname(In NACHWEIS<br>verbleib_nachname<br>eintragen)
verbleib_nachname --> Projekt(In NACHWEIS<br>Projekt eintragen)
Projekt --> VERORTUNG_PUNKT_WKT(In NACHWEIS<br>VERORTUNG_PUNKT_WKT<br>eintragen)
VERORTUNG_PUNKT_WKT --> status_code_lfu(In NACHWEIS<br>status_code_lfu eintragen)
1 --> |6|FUNDORT[Tabelle <br>FUNDORT<br> füllen]
FUNDORT --> TK(In FUNDORT<br>TK eintragen)
TK --> TOPONYM(In FUNDORT<br>TOPONYM eintragen)
TOPONYM --> Hoehe(In FUNDORT<br>Hoehe eintragen)
Hoehe --> LEBENSRAUM(In FUNDORT<br>LEBENSRAUM eintragen)
LEBENSRAUM --> WGS84(In FUNDORT<br>WGS84 eintragen)
WGS84 --> NATURRAUM(In FUNDORT<br>NATURRAUM eintragen)
NATURRAUM --> Landkreis(In FUNDORT<br>Landkreis eintragen)
Landkreis --> naturraum_code_lfu(In FUNDORT<br>naturraum_code_lfu<br>eintragen)
naturraum_code_lfu --> landkreis_code_lfu(In FUNDORT<br>landkreis_code_lfu<br>eintragen)
1 --> |7|SAMMLER[Tabelle <br>SAMMLER<br> fuellen]
-- setting the timestamp
delete from [dbo].[Artcode_LastChange] where Tabelle = 'ArtcodeTemp'
TRUNCATE TABLE [dbo].[Artcode_ArtID];
INSERT INTO [dbo].[Artcode_ArtID](NameID)
select T.ID
FROM ArtcodeTemp AS T
where T.Art_ID IS NULL
-- Setting the Art_ID for missing names
UPDATE T SET T.Art_ID = '9P0#' + REPLICATE('0', 6 - LEN(CAST(A.ID AS varchar))) + CAST(A.ID AS varchar)
-- select T.Art_ID, '9P0#' + REPLICATE('0', 6 - LEN(CAST(A.ID AS varchar))) + CAST(A.ID AS varchar)
FROM ArtcodeTemp AS T INNER JOIN [Artcode_ArtID] A ON T.ID = A.NameId
WHERE T.Art_ID IS NULL
-- Setting the family
TRUNCATE TABLE [dbo].[Artcode_Family]
INSERT INTO [dbo].[Artcode_Family]
([NameID]
,[FamilyNameID]
,[Family])
SELECT
T_0.NameID,
CASE WHEN T_1.TaxonomicRank = 'fam.' THEN T_1.NameID
ELSE
CASE WHEN T_2.TaxonomicRank = 'fam.' AND T_2.IgnoreButKeepForReference = 0 THEN T_2.NameID
ELSE
CASE WHEN T_3.TaxonomicRank = 'fam.' AND T_3.IgnoreButKeepForReference = 0 AND H_2.IgnoreButKeepForReference = 0
THEN T_3.NameID ELSE
CASE WHEN T_4.TaxonomicRank = 'fam.' AND T_4.IgnoreButKeepForReference = 0 AND H_3.IgnoreButKeepForReference = 0
THEN T_4.NameID
ELSE NULL
END END END END AS FamilyNameID,
CASE WHEN T_1.TaxonomicRank = 'fam.' THEN T_1.GenusOrSupragenericName ELSE CASE WHEN T_2.TaxonomicRank = 'fam.' AND
T_2.IgnoreButKeepForReference = 0 THEN T_2.GenusOrSupragenericName ELSE CASE WHEN T_3.TaxonomicRank = 'fam.' AND
T_3.IgnoreButKeepForReference = 0 AND
H_2.IgnoreButKeepForReference = 0 THEN T_3.GenusOrSupragenericName ELSE CASE WHEN T_4.TaxonomicRank = 'fam.' AND
T_4.IgnoreButKeepForReference = 0 AND H_3.IgnoreButKeepForReference = 0 THEN T_4.GenusOrSupragenericName ELSE NULL
END END END END AS Family
FROM dbo.TaxonHierarchy AS H_3 INNER JOIN
dbo.TaxonName AS T_4 ON H_3.NameParentID = T_4.NameID AND H_3.ProjectID = 1128 RIGHT OUTER JOIN
dbo.TaxonName AS T_3 INNER JOIN
dbo.TaxonHierarchy AS H_2 ON T_3.NameID = H_2.NameParentID AND H_2.ProjectID = 1128 ON
H_3.NameID = T_3.NameID RIGHT OUTER JOIN
dbo.TaxonHierarchy AS H_1 INNER JOIN
dbo.TaxonName AS T_2 ON H_1.NameParentID = T_2.NameID AND H_1.ProjectID = 1128 RIGHT OUTER JOIN
dbo.TaxonHierarchy AS H_0 INNER JOIN
dbo.TaxonName AS T_0 ON H_0.NameID = T_0.NameID AND H_0.ProjectID = 1128 INNER JOIN
dbo.TaxonName AS T_1 ON H_0.NameParentID = T_1.NameID AND H_0.ProjectID = 1128 ON H_1.NameID = T_1.NameID AND H_1.ProjectID = 1128 ON
H_2.NameID = T_2.NameID AND H_2.ProjectID = 1128
-- family for synonyms
INSERT INTO [dbo].[Artcode_Family]
([NameID]
,[FamilyNameID]
,[Family])
SELECT S.NameID, F.FamilyNameID, F.Family
FROM [Artcode_Family] F
INNER JOIN dbo.TaxonSynonymy S ON S.SynNameID = F.NameID AND S.ProjectID = 1128
AND NOT EXISTS(SELECT * FROM [Artcode_Family] A WHERE A.NameID = S.NameID)
UPDATE T SET T.fam = UPPER(SUBSTRING(F.Family, 1, 3)), T.id_fam = AF.Art_ID
-- select T.fam, UPPER(SUBSTRING(F.Family, 1, 3)), T.id_fam, AF.Art_ID
FROM ArtcodeTemp AS T INNER JOIN Artcode_Family F ON T.ID = F.NameID
INNER JOIN ArtcodeTemp AS AF ON AF.ID = F.FamilyNameID
--Ausgabesperre, Statistiksperre, Eingabesperre, SAP
UPDATE T SET T.Ausgabesperre = NULL, T.Statistiksperre = NULL, T.Eingabesperre = NULL, T.saP = NULL
-- select T.Ausgabesperre, Statistiksperre, Eingabesperre, SAP
FROM ArtcodeTemp AS T WHERE T.Ausgabesperre = 0
-- setting the rank
UPDATE T SET T.rang = R.rang_id
-- select T.rang, R.rang_id
FROM ArtcodeTemp AS T INNER JOIN [dbo].[Artcode_Rang] R ON R.Code = T.TaxonomicRank
-- setting the rank for synonyms
UPDATE T SET T.rang = 'syn'
-- select T.rang, 'syn'
FROM ArtcodeTemp AS T INNER JOIN TaxonSynonymy S ON T.ID = S.NameID and S.ProjectID = 1128 --and (T.rang is null or T.rang = '')
-- setting the Art_ID_Gueltig for synonyms
UPDATE T SET T.Art_ID_Gueltig = A.Art_ID
-- select T.Art_ID, T.Art_ID_Gueltig, A.Art_ID
FROM ArtcodeTemp AS T INNER JOIN TaxonSynonymy S ON T.ID = S.NameID and S.ProjectID = 1128 AND T.Art_ID_Gueltig IS NULL
INNER JOIN ArtcodeTemp A ON S.SynNameID = A.ID
-- Eintrag Art_ID_Gueltig wo er fehlt
UPDATE A SET A.Art_ID_Gueltig = CASE WHEN E.ExternalNameURI IS NULL THEN NULL ELSE E.ExternalNameURI END
-- SELECT A.Art_ID_Gueltig, CASE WHEN E.ExternalNameURI IS NULL THEN NULL ELSE E.ExternalNameURI END AS Art_ID_Gueltig_E
FROM dbo.TaxonNameExternalID AS E INNER JOIN
dbo.TaxonName AS N ON E.NameID = N.NameID AND E.ExternalDatabaseID = 1003 AND (N.IgnoreButKeepForReference = 0)
INNER JOIN ArtcodeTemp AS A ON A.ID = N.NameID AND A.Art_ID_Gueltig IS NULL
-- setting the Autor_Pos
UPDATE T SET T.Autor_Pos = 1
-- select T.Autor_Pos, 1, T.rang, N.TaxonNameCache, N.TaxonomicRank, ar.DisplayOrder
FROM ArtcodeTemp AS T
inner join TaxonName N on T.id = N.NameID and N.SpeciesEpithet = N.InfraspecificEpithet and N.BasionymAuthors <> ''
inner join Artcode_Rang as AR on N.TaxonomicRank = ar.Code and ar.DisplayOrder < 170
-- setting the Autor_Pos
UPDATE T SET T.Autor_Pos = 2
-- select T.Autor_Pos, 2, T.rang, N.TaxonNameCache, N.TaxonomicRank, ar.DisplayOrder, T.Autor
FROM ArtcodeTemp AS T
inner join TaxonName N on T.id = N.NameID and N.SpeciesEpithet like 'x %' and N.BasionymAuthors <> ''
inner join Artcode_Rang as AR on N.TaxonomicRank = ar.Code and ar.DisplayOrder < 170
-- setting Verantwortung Bayerns
UPDATE T SET T.VeraB = A.AnalysisValue
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 5
-- setting Verantwortung Deutschlands
UPDATE T SET T.VeraD = A.AnalysisValue
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 4
-- setting Gefährdung nach Roter Liste Bayern 2003
UPDATE T SET T.RLB = A.AnalysisValue
-- select T.RLB, A.AnalysisValue
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 1
-- setting Gefährung nach Roter Liste Deutschland 2018
UPDATE T SET T.RLD = A.AnalysisValue
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 62
-- setting Schutzstatus in Bayern
UPDATE T SET T.Schutz_BNatSchG = case when A.AnalysisValue = '§§' then 's' else 'b' end
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 7
UPDATE T SET T.sensu = N.NonNomenclaturalNameSuffix
-- select T.sensu, N.NonNomenclaturalNameSuffix
FROM ArtcodeTemp AS T INNER JOIN [TaxonName] N ON T.ID = N.NameID and N.NonNomenclaturalNameSuffix <> ''
-- setting Schutzstatus in Bayern
UPDATE T SET T.Schutz_BNatSchG = case when A.AnalysisValue = '§§' then 's' else 'b' end
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 7
-- setting id_agg
UPDATE T SET T.id_agg = case when P.Art_ID_Gueltig is null then P.Art_ID else P.Art_ID_Gueltig end
-- select T.id_agg, case when P.Art_ID_Gueltig is null then P.Art_ID else P.Art_ID_Gueltig end
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonHierarchy] H ON T.ID = H.NameID AND H.ProjectID = 1128
inner join ArtcodeTemp P on H.NameParentID = P.ID
inner join dbo.TaxonAcceptedName A on T.id = A.NameID and A.ProjectID = 1128
-- setting id_agg for not accepted names
UPDATE T SET T.id_agg = case when P.Art_ID_Gueltig is null then P.Art_ID else P.Art_ID_Gueltig end
-- select T.id_agg, case when P.Art_ID_Gueltig is null then P.Art_ID else P.Art_ID_Gueltig end
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonHierarchy] H ON T.ID = H.NameID AND H.ProjectID = 1128
inner join ArtcodeTemp P on H.NameParentID = P.ID
WHERE T.id_agg IS NULL OR T.id_agg = ''
-- setting id_agg for not synynyms missing a hierarchy
UPDATE T SET T.id_agg = case when P.Art_ID_Gueltig is null then P.Art_ID else P.Art_ID_Gueltig end
-- select T.id_agg, case when P.Art_ID_Gueltig is null then P.Art_ID else P.Art_ID_Gueltig end
FROM ArtcodeTemp AS T
INNER JOIN [dbo].[TaxonSynonymy] S ON S.NameID = T.ID AND S.ProjectID = 1128
INNER JOIN [dbo].[TaxonHierarchy] H ON S.SynNameID = H.NameID AND H.ProjectID = 1128
inner join ArtcodeTemp P on H.NameParentID = P.ID
WHERE T.id_agg IS NULL OR T.id_agg = ''
-- setting BOLD_ID
UPDATE T SET T.BOLD_ID = E.ExternalNameURI
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameExternalID] E ON T.ID = E.NameID and E.ExternalDatabaseID = 1010
-- setting Bayernstatus
UPDATE T SET T.staby = B.staby
FROM ArtcodeTemp AS T INNER JOIN [dbo].[TaxonNameListAnalysis] A ON T.ID = A.NameID AND A.AnalysisID = 2
INNER JOIN [dbo].[Artcode_Bayernstatus] B ON B.[AnalysisValue] = A.AnalysisValue
INSERT INTO [dbo].[Artcode_LastChange] ([Tabelle])
VALUES ('ArtcodeTemp')
-- setting the timestamp
delete from [dbo].[Artcode_LastChange] where Tabelle = 'Artcode'
TRUNCATE TABLE [dbo].[Artcode]
INSERT INTO Artcode
(Ordnung, Art_ID, Synonym, Art_ID_Gueltig, Gattung, Art, Autor, Autor_Pos, Name_Deutsch, RLB, RLD, FFH_Anh2, FFH_Anh4, FFH_Anh5, VSR_Anh1, Status_IUCN, Schutz_BNatSchG, Ausgabesperre, Statistiksperre,
Eingabesperre, saP, saP_ABC, saP_BC, saP_ZR, rang, sensu, id_agg, staby, VeraB, VeraD, WnASA, EUBrd, BASVO, BOLD_ID, NAME_DG, fam, id_fam, pruefung, ID)
SELECT Ordnung, Art_ID, Synonym, Art_ID_Gueltig, Gattung, Art, Autor, Autor_Pos, Name_Deutsch, RLB, RLD, FFH_Anh2, FFH_Anh4, FFH_Anh5, VSR_Anh1, Status_IUCN, Schutz_BNatSchG, Ausgabesperre, Statistiksperre,
Eingabesperre, saP, saP_ABC, saP_BC, saP_ZR, rang, sensu, id_agg, staby, VeraB, VeraD, WnASA, EUBrd, BASVO, BOLD_ID, NAME_DG, fam, id_fam, pruefung, ID
FROM ArtcodeTemp
INSERT INTO [dbo].[Artcode_LastChange] ([Tabelle])
VALUES ('Artcode')Nach einem Click auf den Button Export Artcode im Hauptformular öffnet sich ein Fenster wie unten dargestellt.
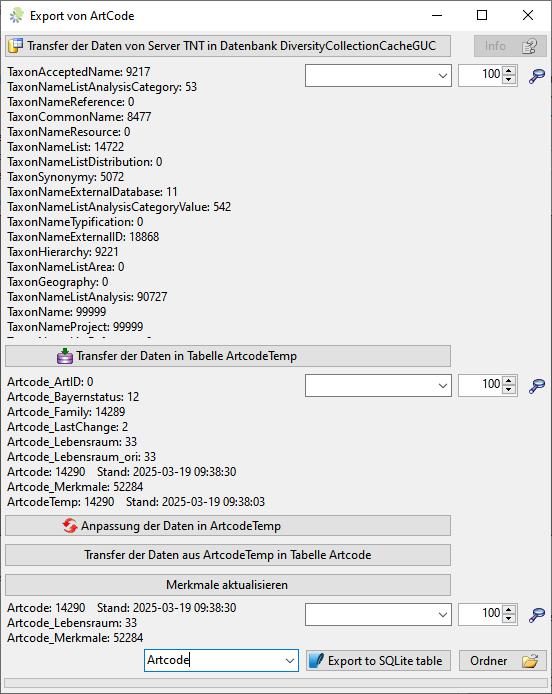
Mit den 
graph TD;
DTN[<i class="fa-fw fas fa-database"></i> DTN_Plants<br><br><i class="fa-fw fas fa-table"></i> TaxonName<br><i class="fa-fw fas fa-table"></i> ...]
GUC[<i class="fa-fw fas fa-database"></i> DC_CacheGUC<br><br><i class="fa-fw fas fa-table"></i> TaxonName<br><i class="fa-fw fas fa-table"></i> ...]
DTN ==>|<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_TransferTaxa</b>| GUC
GUC_Artcode(<i class="fa-fw fas fa-database"></i> DC_CacheGUC<br><br><i class="fa-fw fas fa-table"></i> TaxonName<br><i class="fa-fw fas fa-table"></i> ...<br><i class="fa-fw fas fa-long-arrow-alt-down"></i><br><i class="fa-fw fas fa-eye"></i> GUC_Artcode)
GUC -.- GUC_Artcode
GUC_ArtcodeTemp(<i class="fa-fw fas fa-database"></i> DC_CacheGUC<br><br><i class="fa-fw fas fa-table"></i> ArtcodeTemp)
GUC_ArtcodeTempAngepasst(<i class="fa-fw fas fa-table"></i> ArtcodeTemp)
GUC_Artcode -->|<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_TransferInArtcode</b>| GUC_ArtcodeTemp
GUC_ArtcodeTemp -.-o|<i class="fa-fw fas fa-edit"></i> Prozedur<br><b>GUC_AnpassungArtcodeTemp</b>| GUC_ArtcodeTempAngepasst
Artcode(<i class="fa-fw fas fa-table"></i> Artcode)
GUC_ArtcodeTempAngepasst -->|<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_TransferArtcodeTempInArtcode</b>| Artcode
Merkmale[<i class="fa-fw fas fa-table"></i> Artcode_Merkmale]
TaxonNameListAnalysis[<i class="fa-fw fas fa-table"></i> TaxonNameListAnalysis]
DTN --> TaxonNameListAnalysis
TaxonNameListAnalysis --> |<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_Transfer_Artcode_Merkmale</b>| Merkmale
Artcode --> SQLite[<i class="fa-fw fas fa-database"></i> SQLite]
Merkmale --> SQLite[<i class="fa-fw fas fa-database"></i> SQLite]
Lebensraum[<i class="fa-fw fas fa-table"></i> Artcode_Lebensraum] --> SQLite[<i class="fa-fw fas fa-database"></i> SQLite]
In der Regel werden folgende Tabellen werden benötigt:
Mit einem Click auf den Button kann man das Verzeichnis in dem die 
| Column | Data type | Description | Nullable | Relation |
|---|---|---|---|---|
| Ordnung | nvarchar (3) | entählt immer den Wert 9P0 entsprechend dem Code der GUC | NO | - |
| Art_ID | nvarchar (12) | Code der GUC | YES | - |
| Synonym | bit | Ob der Name ein Synonym ist | YES | - |
| Art_ID_Gueltig | nvarchar (12) | GUD ID der Gültigen Art in Fall eines Synonyms, falls gültiger Name, dann die Art_ID | YES | - |
| Gattung | nvarchar (255) | Die Gattung | YES | - |
| Art | nvarchar (255) | für Taxa unterhalb der Gattung. Das Epitheton der Art. Im Fall e.g. einer anderen Taxonomischen Stufe alles Gattung und den Authoren | YES | - |
| Autor | nvarchar (255) | die Authoren | YES | - |
| Autor_Pos | tinyint | Position des Authors innerhalb des Textes im Feld Art. Falls kein Author dann NULL | YES | - |
| Name_Deutsch | nvarchar (255) | Der aktuelle deutsche Namen des Taxon | YES | - |
| RLB | nvarchar (5) | Gefährdung nach Roter Liste Bayern 2024. NB für den Wert “Karo” und langem Bindestrich | YES | - |
| RLD | nvarchar (5) | Gefährdung nach Roter Liste Deutschland 2018 | YES | - |
| FFH_Anh2 | nvarchar (1) | - | YES | - |
| FFH_Anh4 | nvarchar (1) | - | YES | - |
| FFH_Anh5 | nvarchar (1) | - | YES | - |
| VSR_Anh1 | nvarchar (1) | - | YES | - |
| Status_IUCN | nvarchar (12) | - | YES | - |
| Schutz_BNatSchG | nvarchar (12) | Schutzsstatus in Bayern. Übersetzung von “§§” in “s” sonst “b” | YES | - |
| Ausgabesperre | bit | immer 0 | NO | - |
| Statistiksperre | bit | immer 0 | NO | - |
| Eingabesperre | bit | immer 0 | NO | - |
| saP | bit | immer 0 | NO | - |
| saP_ABC | bit | immer 0 | NO | - |
| saP_BC | bit | immer 0 | NO | - |
| saP_ZR | bit | immer 0 | NO | - |
| rang | nvarchar (5) | Taxonomischer Rang | YES | - |
| sensu | nvarchar (255) | NonNomenclaturalNameSuffix mit diversen Anpassungen für die GUC | YES | - |
| id_agg | nvarchar (10) | die Art_ID der GUC des übergeordneten gültigen Taxon | YES | - |
| staby | nvarchar (1) | Status in Bayern | YES | - |
| VeraB | nvarchar (1) | Verantwortung Bayerns nach RL Bayern 2003 | YES | - |
| VeraD | nvarchar (4) | Verantwortung Deutschlands nach RL Bayern 2003 | YES | - |
| WnASA | nvarchar (4) | - | YES | - |
| EUBrd | bit | - | NO | - |
| BASVO | nvarchar (1) | - | YES | - |
| BOLD_ID | int | DNA barcode ID collected by the Barcode of Life Datasystems (BOLD) | YES | - |
| NAME_DG | nvarchar (255) | - | YES | - |
| fam | nvarchar (255) | 3 Buchstaben Code der Familie der GUC | YES | - |
| id_fam | nvarchar (255) | Art_ID der Familie | YES | - |
| pruefung | tinyint | - | YES | - |
| ID | int | NameID aus DTN | NO | - |
| Column | Data type | Description | Nullable | Relation |
|---|---|---|---|---|
| Art_ID | nvarchar (10) | Code der GUC | NO | - |
| Merkmal | nvarchar (5) | 5 stelliger Code der GUC für Merkmale | NO | - |
| Wert | nvarchar (255) | Wert des Merkmals | NO | - |
| Code | Descripton |
|---|---|
| TAXNR | NameID aus DTN |
| ST9P0 | Floristischer Status |
| NEOBI | N für Floristischen Status ‘E’, ‘E?’, ‘P’, ‘T’, ‘T?’, ‘U’ |
| RL_A | Gefährdung n. RL Bayern 2024 - Alpen |
| RL_HB | Gefährdung n. RL Bayern 2024 - Hügel- u. Bergland |
| Column | Data type | Description | Nullable | Relation |
|---|---|---|---|---|
| Art_ID | nvarchar (10) | Code der GUC | NO | - |
| lr_id | int | Code der GUC für Lebensraum | NO | - |
| vk_id | tinyint | Code des Vorkommens analog Tabelle | NO | - |
| Code | Descripton |
|---|---|
| 1 | Hauptvorkommen |
| 2 | Vorkommen |
| 3 | potentielles Vorkommen |
| 4 | Jagdhabitat |
| Code | Descripton |
|---|---|
| 11 | Alpine Felsen, Schutthalden, Gletscher, Firn |
| 12 | Alpine Rasen, Wiesen, Weiden und Staudenfluren |
| 13 | Alpine Zwergstrauchheiden und Krummholz |
| 14 | Alpine Wälder |
| 21 | Quellen |
| 22 | Fließgewässer |
| 23 | Stillgewässer |
| 31 | Moore und Sümpfe |
| 32 | Feucht- und Nassgrünland, feuchte Hochstaudenfluren |
| 41 | Magerrasen, Heiden, Säume und wärmeliebende Gebüsche |
| 42 | Rohböden, Abgrabungen |
| 43 | Felsen, Schuttfluren, Blockhalden |
| 44 | Weinberge |
| 51 | Hecken, Feldgehölze, Gebüsche, Parkanlagen, Baumgruppen, Einzelbäume |
| 52 | Streuobstbestände |
| 61 | Nadelwälder auf mittleren Standorten |
| 62 | Laub- und Mischwälder auf mittleren Standorten |
| 63 | Nass- und Feuchtwälder |
| 64 | Trockenwälder |
| 71 | Grünland mittlerer Standorte |
| 72 | Äcker, Sonderkulturen und Raine |
| 81 | Böschungen, Dämme, Bahngelände und andere Verkehrsflächen |
| 82 | Höhlen, Stollen und Keller |
| 83 | Siedlungen, Gebäude und andere urbane Lebensräume |
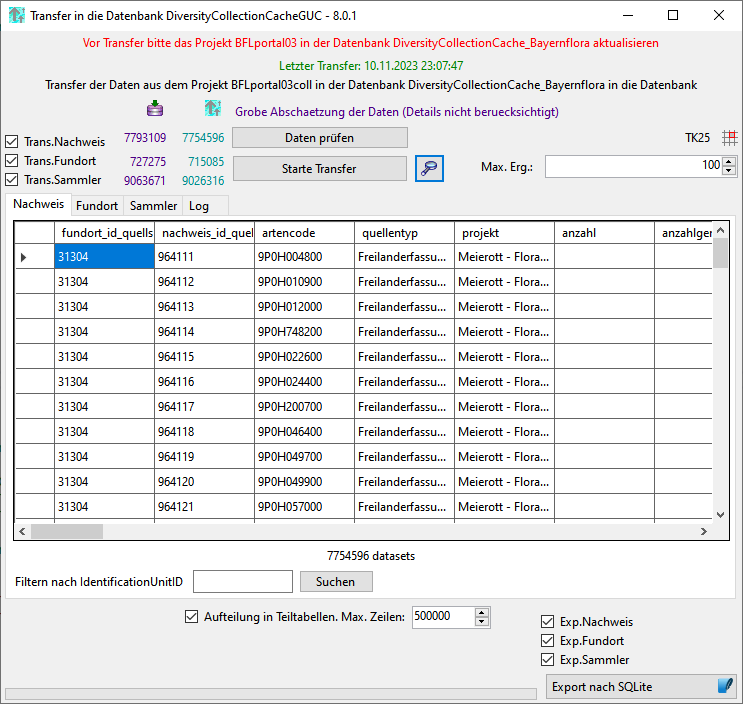
Beim Start des Programms werden die aktuellen Zahlen in den Quellen und dem Cache für die GUC geladen. In der Kopfzeile werden das Datum des letzten Transfers für das Projekt BFLportal03coll sowie die Artcodes angezeigt. Beide dienen als Grundlage für die Daten die an die GUC geliefert werden und sollten entsprechend aktuell sein.
--- title: Quellen (Server, Datenbanken) für die Daten --- graph LR; Tnt[<i class="fa-fw fas fa-server"></i> Server<br>TNT] -->DTN_Plants(<i class="fa-fw fas fa-database"></i> Database<br>DTN_Plants)-->DTN_Plants_Bfl(<i class="fa-fw fas fa-lock"></i> Projekt<br>BFLnames) Bfl[<i class="fa-fw fas fa-server"></i> Server<br>BFL] -->DC_Bfl(<i class="fa-fw fas fa-database"></i> Database<br>DC_Bayernflora)-->DC_Bfl_3(<i class="fa-fw fas fa-lock"></i> Projekt<br>BFLportal03coll) DC_Bfl_3-->DC_Bfl_Cache(<i class="fa-fw fas fa-database"></i> Database<br>DC_BayernfloraCache) DTN_Plants_Bfl-->DC_Bfl_Cache(<i class="fa-fw fas fa-database"></i> Database<br>DC_BayernfloraCache) DC_Bfl_Cache-->DC_Bfl_Guc_Cache(<i class="fa-fw fas fa-database"></i> Database<br>DC_BayernfloraGucCache)
Vor dem Export für GUC müssen die Daten der hierfür relevanten Projekte in der DiversityCollectionCache_BayernFlora aktualisiert werden.
Vor dem Export für GUC müssen die Daten in der Tabelle Artcode aktualisiert werden.
Mit einem Click auf den Button wird die Prozedur TransferToGUC (s. unten) gestartet. Diese Überträgt die Daten aus der Datenbank DiversityCollectionCache_BayernFlora in Abhängigkeit der ausgewählten Optionen in die Tabellen der Datenbank DiversityCollectionCacheGUC und bereitet diese für den Export auf.
--- title: PROCEDURE TransferToGUC --- graph TD; TransferToGUC[<i class="fa-fw fas fa-arrows-rotate"></i> Hauptprozedur<br>zum Übertrag] -->Festhalten_Zeitpunkts(<i class="fa-fw fas fa-stopwatch"></i> Festhalten<br>des Zeitpunkts)-->Festhalten_Zeitpunkte(<i class="fa-fw fas fa-stopwatch"></i> Festhalten<br>des Zeitpunkts<br>aller Schritte<br>→Tabelle _Log)-->fundort_datum[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen der Daten in<br>_fundort_letzte_Aend...<br>→Tabelle _Log]-->Alle_Zeitpunkte[<i class="fa-fw fas fa-stopwatch"></i> ...<br>→Tabelle _Log] Festhalten_Zeitpunkts-->LoeschenTabellen(<i class="fa-fw fas fa-trash"></i> Löschen<br>der Tabellen:<br>_Status, <br>NACHWEIS, <br>FUNDORT, <br>SAMMLER) -->Status[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen der Daten<br>in _Status] LoeschenTabellen-->_Identification[<i class="fa-fw fas fa-table"></i> Tabelle<br>_Identification] -->Loeschen_Identification(<i class="fa-fw fas fa-trash"></i> Löschen<br>der Daten)-->Einlesen_Identification[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten ]-->Loeschen_Identification_ohne_artencode(<i class="fa-fw fas fa-trash"></i> Entfernung<br>von Daten<br>die keinen<br>artencode haben) Loeschen_Identification_ohne_artencode-->Einlesen_Nachweis LoeschenTabellen-->Nachweis[<i class="fa-fw fas fa-table"></i> Tabelle<br>NACHWEIS]-->Einlesen_Nachweis[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten]-->Loeschen_Nachweis_ohne_Jahr(<i class="fa-fw fas fa-trash"></i>Entfernung<br>von Daten<br>die kein<br>Jahr haben)-->Eintag_Nachweis_Status(<i class="fa-fw fas fa-pen"></i> Eintrag<br>des Status)-->Eintag_Nachweis_quellentyp(<i class="fa-fw fas fa-pen"></i> Eintrag<br>des Quellentyp)-->Eintag_Nachweis_verbleib_nachname(<i class="fa-fw fas fa-pen"></i> Eintrag<br>verbleib_nachname)-->Eintag_Nachweis_projekt(<i class="fa-fw fas fa-pen"></i> Eintrag<br>projekt)-->Eintag_Nachweis_VERORTUNG_PUNKT_WKT(<i class="fa-fw fas fa-pen"></i> Eintrag<br>VERORTUNG_PUNKT_WKT)-->Eintag_Nachweis_status_code_lfu(<i class="fa-fw fas fa-pen"></i> Eintrag<br>status_code_lfu)-->Export_Nachweis(<i class="fa-fw fas fa-download"></i>) Status-->Eintag_Nachweis_Status LoeschenTabellen-->FUNDORT[<i class="fa-fw fas fa-table"></i> Tabelle<br>FUNDORT]-->Einlesen_FUNDORT[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten]-->Eintag_FUNDORT_tk(<i class="fa-fw fas fa-pen"></i> Eintrag<br>TK, Quadrant, ...)-->Eintag_FUNDORT_toponym(<i class="fa-fw fas fa-pen"></i> Eintrag<br>toponym)-->Eintag_FUNDORT_Hoehe(<i class="fa-fw fas fa-pen"></i> Eintrag<br>Hoehe)-->Eintag_FUNDORT_lebensraum(<i class="fa-fw fas fa-pen"></i> Eintrag<br>lebensraum)-->Eintag_FUNDORT_WGS84(<i class="fa-fw fas fa-pen"></i> Eintrag<br>WGS84)-->Eintag_FUNDORT_naturraum(<i class="fa-fw fas fa-pen"></i> Eintrag<br>naturraum)-->Eintag_FUNDORT_landkreis(<i class="fa-fw fas fa-pen"></i> Eintrag<br>landkreis)-->Eintag_FUNDORT_naturraum_code_lfu(<i class="fa-fw fas fa-pen"></i> Eintrag<br>naturraum_code_lfu)-->Eintag_FUNDORT_landkreis_code_lfu(<i class="fa-fw fas fa-pen"></i> Eintrag<br>landkreis_code_lfu)-->Export_Fundort(<i class="fa-fw fas fa-download"></i>) LoeschenTabellen-->SAMMLER[<i class="fa-fw fas fa-table"></i> Tabelle<br>SAMMLER]-->Einlesen_SAMMLER[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Einlesen<br>der Daten]-->Export_Sammler(<i class="fa-fw fas fa-download"></i>)
Der Zeitbedarf sowie die Zahl der betroffenen Datensätze wird im Log angezeigt
Um die Daten als SQLite zu exportieren einfach auf den Export nach SQLite 
Nach einem Click auf den Button TK25 
Alle Objekte für diese Funktion befinden sich in der Datenbank im Schema TK25.
Ein Click auf den Button Start Transfer startet die Prozedur TK25.TransferToQuadrant mit den ausgewählten Parameter für das Jahr und den tax. Rang. Diese Prozedur füllt die Tabelle TK25.Quadrant und TK25.Identification.
Alle Schritte werden in die Tabelle _Log eingetragen und können mit dem Button 
---
title: PROCEDURE TK25.TransferToQuadrant
---
graph TD;
Alle_Zeitpunkte[<i class="fa-fw fas fa-stopwatch"></i> ...<br>→Tabelle<br><i class="fa-fw fas fa-table"></i> _Log]
TNT[<i class="fa-fw fas fa-server"></i> TNT<br><i class="fa-fw fas fa-database"></i> DTN_Plants<br>]
Cache[<i class="fa-fw fas fa-server"></i> BFL<br><i class="fa-fw fas fa-database"></i> DC_Cache_BFL<br><i class="fa-fw fas fa-lock"></i> BFLportal02coll<br>]
Clear[<i class="fa-fw fas fa-trash"></i> Löschen<br>der Tabellen<br><u>TK25.Identification</u><br><u>TK25.Quadrant</u>]
Identification[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Füllen der Tabelle<br><u>TK25.Identification</u>]
Clear --> Identification
Cache --> Identification
Identification --> former
former(<i class="fa-fw fas fa-trash"></i> Löschen<br>von Bestimmungen<br>vor gewähltem Jahr<br>in Tabelle<br><u>TK25.Identification</u>)
former --> accepted
TNT --> accepted
accepted(<i class="fa-fw fas fa-edit"></i> Eintrag<br>der ID von<br>Akzeptierten Namen<br>in Tabelle<br><u>TK25.Identification</u>)
accepted --> higher
TNT --> higher
higher(<i class="fa-fw fas fa-edit"></i> Eintrag<br>der ID<br>des höheren Taxons<br>in Tabelle<br><u>TK25.Identification</u>)
higher --> Quadrant
Cache --> Quadrant
Quadrant[<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Füllen<br>von Tabelle <br><u>TK25.Quadrant</u>]
Export[<i class="fa-fw fas fa-file"></i> Export<br>in Datei<br>TK25export.txt]
Quadrant --> Export
Um die Daten als Textdatei zu exportieren auf den Export Button clicken. Dadurch wird im Projektverzeichnis …/DiversityWorkbench/GUC eine Datei mit dem Name TK25export.txt erstellt.