GUC
Artcode
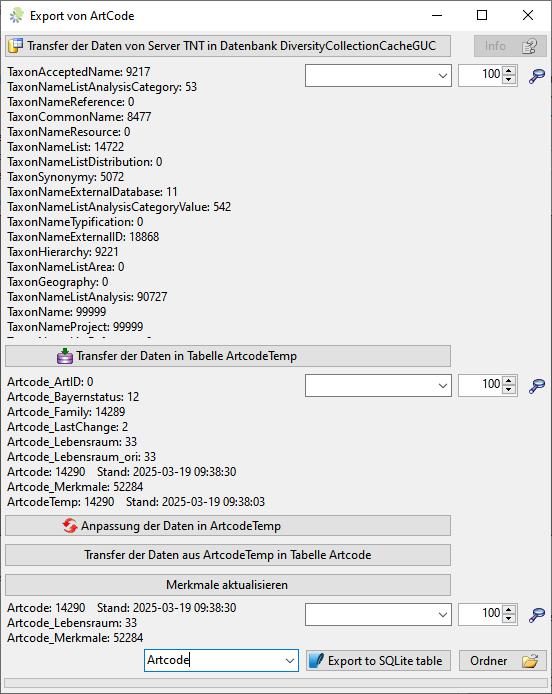
Nach einem Click auf den Button Export Artcode im Hauptformular öffnet sich ein Fenster wie unten dargestellt.
Transfer der Daten aus DTN
Mit den 
graph TD;
DTN[<i class="fa-fw fas fa-database"></i> DTN_Plants<br><br><i class="fa-fw fas fa-table"></i> TaxonName<br><i class="fa-fw fas fa-table"></i> ...]
GUC[<i class="fa-fw fas fa-database"></i> DC_CacheGUC<br><br><i class="fa-fw fas fa-table"></i> TaxonName<br><i class="fa-fw fas fa-table"></i> ...]
DTN ==>|<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_TransferTaxa</b>| GUC
GUC_Artcode(<i class="fa-fw fas fa-database"></i> DC_CacheGUC<br><br><i class="fa-fw fas fa-table"></i> TaxonName<br><i class="fa-fw fas fa-table"></i> ...<br><i class="fa-fw fas fa-long-arrow-alt-down"></i><br><i class="fa-fw fas fa-eye"></i> GUC_Artcode)
GUC -.- GUC_Artcode
GUC_ArtcodeTemp(<i class="fa-fw fas fa-database"></i> DC_CacheGUC<br><br><i class="fa-fw fas fa-table"></i> ArtcodeTemp)
GUC_ArtcodeTempAngepasst(<i class="fa-fw fas fa-table"></i> ArtcodeTemp)
GUC_Artcode -->|<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_TransferInArtcode</b>| GUC_ArtcodeTemp
GUC_ArtcodeTemp -.-o|<i class="fa-fw fas fa-edit"></i> Prozedur<br><b>GUC_AnpassungArtcodeTemp</b>| GUC_ArtcodeTempAngepasst
Artcode(<i class="fa-fw fas fa-table"></i> Artcode)
GUC_ArtcodeTempAngepasst -->|<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_TransferArtcodeTempInArtcode</b>| Artcode
Merkmale[<i class="fa-fw fas fa-table"></i> Artcode_Merkmale]
TaxonNameListAnalysis[<i class="fa-fw fas fa-table"></i> TaxonNameListAnalysis]
DTN --> TaxonNameListAnalysis
TaxonNameListAnalysis --> |<i class="fa-fw fas fa-arrow-alt-circle-down"></i> Prozedur<br><b>GUC_Transfer_Artcode_Merkmale</b>| Merkmale
Artcode --> SQLite[<i class="fa-fw fas fa-database"></i> SQLite]
Merkmale --> SQLite[<i class="fa-fw fas fa-database"></i> SQLite]
Lebensraum[<i class="fa-fw fas fa-table"></i> Artcode_Lebensraum] --> SQLite[<i class="fa-fw fas fa-database"></i> SQLite]
Schritt 1
- Mit einem Click auf den Button werden die Daten aus der Datenbank DiversityTaxonNames_Plants auf dem tnt.diversityworkbench.de in gleich aufgebaute Tabellen in der Datenbank DiversityCollectionCacheGUC auf dem bfl.diversityworkbench.de übertragen.
- Die Zahl der Datensätze in den Tabellen werden wie in der Abbildung oben gelistet
- Der Transfer erflolgt durch die stored procedure GUC_TransferTaxa in DiversityCollectionCacheGUC.
- Diese löscht den Inhalt der Taxon Tabellen in DiversityCollectionCacheGUC und befüllt die Tabellen mit dem aktuellen Inhalt aus DiversityTaxonNames_Plants
Schritt 2
- Mit einem Click auf den Button wird die Prozedur GUC_TransferInArtcode gestartet: Auslesen der Daten aus Sicht GUC_Artcode und import in Tabelle ArtcodeTemp
Schritt 3
- Mit einem Click auf den Button wird die Prozedur GUC_AnpassungArtcodeTemp gestarted. Diese überarbeitet die Daten in Tabelle ArtcodeTemp um sie an die Anforderungen von GUC anzupassen.
Schritt 4
- Übertrag der Daten aus ArtcodeTemp in Artcode erfolgt Mit einem Click auf den Button nach Schritt 3
Schritt 5
- Mit einem Click auf den Button wird die Prozedur GUC_Transfer_Artcode_Merkmale gestartet: Auslesen der Daten aus Tabelle TaxonNameListAnalysis und import in Tabelle Artcode_Merkmale sowie Korrekturen der Ausgabe.
Schritt 6
- Auswahl einer zu exportierenden Tabelle und Export der Daten in SQLite Datenbank mit click auf dem button.
In der Regel werden folgende Tabellen werden benötigt:
- Artcode
- Artcode_Lebensraum
- Artcode_Merkmale
Öffnen der SQLite Datenbank
Mit einem Click auf den Button kann man das Verzeichnis in dem die 
Tabellen
Table Artcode
| Column | Data type | Description | Nullable | Relation |
|---|---|---|---|---|
| Ordnung | nvarchar (3) | entählt immer den Wert 9P0 entsprechend dem Code der GUC | NO | - |
| Art_ID | nvarchar (12) | Code der GUC | YES | - |
| Synonym | bit | Ob der Name ein Synonym ist | YES | - |
| Art_ID_Gueltig | nvarchar (12) | GUD ID der Gültigen Art in Fall eines Synonyms, falls gültiger Name, dann die Art_ID | YES | - |
| Gattung | nvarchar (255) | Die Gattung | YES | - |
| Art | nvarchar (255) | für Taxa unterhalb der Gattung. Das Epitheton der Art. Im Fall e.g. einer anderen Taxonomischen Stufe alles Gattung und den Authoren | YES | - |
| Autor | nvarchar (255) | die Authoren | YES | - |
| Autor_Pos | tinyint | Position des Authors innerhalb des Textes im Feld Art. Falls kein Author dann NULL | YES | - |
| Name_Deutsch | nvarchar (255) | Der aktuelle deutsche Namen des Taxon | YES | - |
| RLB | nvarchar (5) | Gefährdung nach Roter Liste Bayern 2024. NB für den Wert “Karo” und langem Bindestrich | YES | - |
| RLD | nvarchar (5) | Gefährdung nach Roter Liste Deutschland 2018 | YES | - |
| FFH_Anh2 | nvarchar (1) | - | YES | - |
| FFH_Anh4 | nvarchar (1) | - | YES | - |
| FFH_Anh5 | nvarchar (1) | - | YES | - |
| VSR_Anh1 | nvarchar (1) | - | YES | - |
| Status_IUCN | nvarchar (12) | - | YES | - |
| Schutz_BNatSchG | nvarchar (12) | Schutzsstatus in Bayern. Übersetzung von “§§” in “s” sonst “b” | YES | - |
| Ausgabesperre | bit | immer 0 | NO | - |
| Statistiksperre | bit | immer 0 | NO | - |
| Eingabesperre | bit | immer 0 | NO | - |
| saP | bit | immer 0 | NO | - |
| saP_ABC | bit | immer 0 | NO | - |
| saP_BC | bit | immer 0 | NO | - |
| saP_ZR | bit | immer 0 | NO | - |
| rang | nvarchar (5) | Taxonomischer Rang | YES | - |
| sensu | nvarchar (255) | NonNomenclaturalNameSuffix mit diversen Anpassungen für die GUC | YES | - |
| id_agg | nvarchar (10) | die Art_ID der GUC des übergeordneten gültigen Taxon | YES | - |
| staby | nvarchar (1) | Status in Bayern | YES | - |
| VeraB | nvarchar (1) | Verantwortung Bayerns nach RL Bayern 2003 | YES | - |
| VeraD | nvarchar (4) | Verantwortung Deutschlands nach RL Bayern 2003 | YES | - |
| WnASA | nvarchar (4) | - | YES | - |
| EUBrd | bit | - | NO | - |
| BASVO | nvarchar (1) | - | YES | - |
| BOLD_ID | int | DNA barcode ID collected by the Barcode of Life Datasystems (BOLD) | YES | - |
| NAME_DG | nvarchar (255) | - | YES | - |
| fam | nvarchar (255) | 3 Buchstaben Code der Familie der GUC | YES | - |
| id_fam | nvarchar (255) | Art_ID der Familie | YES | - |
| pruefung | tinyint | - | YES | - |
| ID | int | NameID aus DTN | NO | - |
Table Artcode_Merkmale
| Column | Data type | Description | Nullable | Relation |
|---|---|---|---|---|
| Art_ID | nvarchar (10) | Code der GUC | NO | - |
| Merkmal | nvarchar (5) | 5 stelliger Code der GUC für Merkmale | NO | - |
| Wert | nvarchar (255) | Wert des Merkmals | NO | - |
Codes für Merkmal
| Code | Descripton |
|---|---|
| TAXNR | NameID aus DTN |
| ST9P0 | Floristischer Status |
| NEOBI | N für Floristischen Status ‘E’, ‘E?’, ‘P’, ‘T’, ‘T?’, ‘U’ |
| RL_A | Gefährdung n. RL Bayern 2024 - Alpen |
| RL_HB | Gefährdung n. RL Bayern 2024 - Hügel- u. Bergland |
Table Artcode_Lebensraum
| Column | Data type | Description | Nullable | Relation |
|---|---|---|---|---|
| Art_ID | nvarchar (10) | Code der GUC | NO | - |
| lr_id | int | Code der GUC für Lebensraum | NO | - |
| vk_id | tinyint | Code des Vorkommens analog Tabelle | NO | - |
Codes für Vorkommen
| Code | Descripton |
|---|---|
| 1 | Hauptvorkommen |
| 2 | Vorkommen |
| 3 | potentielles Vorkommen |
| 4 | Jagdhabitat |
Codes für Lebensraum
| Code | Descripton |
|---|---|
| 11 | Alpine Felsen, Schutthalden, Gletscher, Firn |
| 12 | Alpine Rasen, Wiesen, Weiden und Staudenfluren |
| 13 | Alpine Zwergstrauchheiden und Krummholz |
| 14 | Alpine Wälder |
| 21 | Quellen |
| 22 | Fließgewässer |
| 23 | Stillgewässer |
| 31 | Moore und Sümpfe |
| 32 | Feucht- und Nassgrünland, feuchte Hochstaudenfluren |
| 41 | Magerrasen, Heiden, Säume und wärmeliebende Gebüsche |
| 42 | Rohböden, Abgrabungen |
| 43 | Felsen, Schuttfluren, Blockhalden |
| 44 | Weinberge |
| 51 | Hecken, Feldgehölze, Gebüsche, Parkanlagen, Baumgruppen, Einzelbäume |
| 52 | Streuobstbestände |
| 61 | Nadelwälder auf mittleren Standorten |
| 62 | Laub- und Mischwälder auf mittleren Standorten |
| 63 | Nass- und Feuchtwälder |
| 64 | Trockenwälder |
| 71 | Grünland mittlerer Standorte |
| 72 | Äcker, Sonderkulturen und Raine |
| 81 | Böschungen, Dämme, Bahngelände und andere Verkehrsflächen |
| 82 | Höhlen, Stollen und Keller |
| 83 | Siedlungen, Gebäude und andere urbane Lebensräume |

 Transfer der Daten … in …GUC
Transfer der Daten … in …GUC Transfer der Daten in Tabelle ArtcodeTemp
Transfer der Daten in Tabelle ArtcodeTemp Anpassung der Daten in ArtcodeTemp
Anpassung der Daten in ArtcodeTemp